Profesionales
DIRECTORA DEL INBIOMED
La UCE realiza pruebas de PCR con conocimiento de causa
'Los pequeños detalles, hacen grandes diferencias', ha dicho Baldeón
Lunes, 13 de julio de 2020, a las 10:24

Lucy Baldeón, directora del Inbiomed. |
Redacción. Quito
Tras la publicación de la nota ‘Nueva polémica con las pruebas CoVID19 del Municipio de Quito: la UCE determinaba las muestras solo con un gen’, la directora del Instituto de Biomedicina (Inbiomed), Lucy Baldeón, ha pedido un espacio para explicar el trabajo realizado en el procesamiento de las pruebas para CoVID19.
“Compartimos esta aclaración técnica del procesamiento de los kits de PCR en tiempo real, para que se conozca el arduo trabajo que se realiza en los diferentes laboratorios que apoyan en el diagnóstico de CoVID19 y primordialmente para que la ciudadanía conozca que en el Inbiomed-UCE, lo realizamos con estándares profesionales de alto nivel”, ha señalado Baldeón.
A continuación el texto remitido a EDICIÓN MÉDICA.
En el Inbiomed contamos con profesionales capacitados en el campo de la Medicina, Biología Molecular, Inmunología y Virología. Dichos profesionales poseemos amplia experiencia en la técnica de PCR en tiempo real por lo tanto hemos realizado rigurosos protocolos de estandarización y calibración del kit Isopollo, los cuales detallamos a continuación:
Recibimos por parte del Municipio de Quito, un kit de RT-PCR LAMP (Loop-mediated isothermal amplification), el cual es una reacción de tipo PCR (Reacción en Cadena de la Polimerasa), en la cual una enzima polimerasa sintetiza nuevas cadenas de ADN a partir de un molde de ARN, sin la utilización de ciclos térmicos como en una PCR convencional, sino a través de un solo ciclo de temperatura continua, obteniendo resultados en menor tiempo que con las reacciones comunes. Para la visualización en tiempo real de la amplificación de ADN, se usa un fluorocromo intercalante que emite fluorescencia al ligarse con las cadenas dobles de ADN, así, a mayor cantidad de ADN, mayor es la fluorescencia captada por los termocicladores de tiempo real.
El kit Isopollo® COVID-19, fue desarrollado para la amplificación de dos genes en reacciones de RT-PCR LAMP separadas: 1) Gen no estructural RdRp (Primer CR) que se lee con el fluorócromo FAM y 2) Gen estructural N (Primer CN) que se lee con el fluorócromo FAM. Adicionalmente tiene un control positivo que es amplificado con su propio juego de primers independientes del CR y/o CN.
Para confirmar los resultados positivos y negativos, usamos los siguientes controles:
- Control negativo gen RdRp: Mix con el primer CR + Agua destilada (propia del kit).
- Control negativo gen N: Mix con el primer CN + Agua destilada (propia del kit).
- Control positivo kit: Mix individual con el Control Primer + Control template
- Control positivo gen RdRp de campo*: Mix con el primer CR + ARN 61.1
- Control positivo gen N de campo*: Mix con el primer CN + ARN 61.1
Control de campo* son muestras de ARN de pacientes que fueron previamente diagnosticados con COVID-19 en un laboratorio externo.
Las reacciones de PCR son válidas solo si existe amplificación positiva para los controles positivos de campo y del kit, y amplificación negativa para los controles negativos. Una curva de amplificación positiva está compuesta de las siguientes fases: línea base, crecimiento exponencial, crecimiento linear y meseta (plató). El Cycle threshold (CT) debe estar ubicado dentro de los 40 ciclos de amplificación (según instrucciones del fabricante). Un resultado negativo se determina cuando no existen curvas de amplificación, o el CT es superior a 40.
Adicionalmente validamos cada reacción de amplificación mediante el análisis de la curva de disociación (curvas melting), en donde se captura la fluorescencia que va disminuyendo inversamente proporcional al aumento de temperatura programada para este paso de validación. De esta forma se puede predecir el tamaño de los productos de PCR con relación al control positivo de campo (ARN positivo para COVID-19), y se concluye que la curva de amplificación corresponde a un fragmente del genoma viral y no a productos inespecíficos o dímeros que pueden generar fluorescencia.
Validación de Sensibilidad y Especificidad del Kit.
Nuestro laboratorio no ha podido realizar validación de la sensibilidad de este kit debido varias limitaciones técnicas como son: 1. Límite de detección (no realizado): A través de ingeniería genética se puede generar fragmentos de genomas virales in-vitro para utilizarlos como estándares de validación. Para esto se necesita generar una curva estándar con fragmentos de ARN viral con tamaños y concentraciones conocidas:
a. Plásmidos purificados con fragmentos de genoma viral insertado.
b. Plásmidos clonados con fragmentos de genoma viral insertado y dentro de células
competentes (Ejm. E. coli).
c. Genomas virales de SARS-CoV-2 purificados y cuantificados.
2. Límite de cuantificación (no realizado): Idem al literal 1.
Validación de especificidad
No fue realizada debido a que se necesitan varios aislados virales para garantizar que no existan reacciones cruzadas con otros patógenos respiratorios. Inbiomed, no se cuenta con un biobanco serológico de virus disponible para este efecto.
A pesar de las limitaciones técnicas anteriormente explicadas, para conocer las características de este kit, realizamos los ensayos de concordancia y de eficiencia de primers.
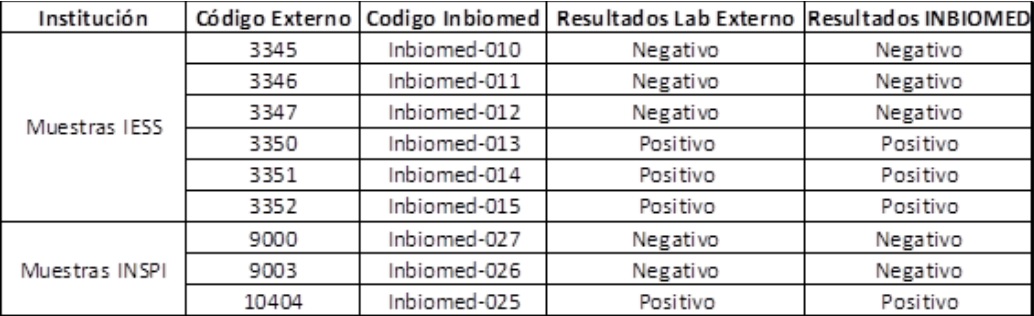
A inicios del mes de mayo, solicitamos a dos laboratorios externos (INSPI e IESS-HCAM) muestras de pacientes positivos y negativos que fueron diagnosticados en sus respectivos laboratorios con técnicas de PCR en tiempo real. Estos test se realizaron en INBIOMED sin que nosotros conozcamos previamente los resultados; los cuales fueron corroborados posteriormente con los responsables de los 2 laboratorios.
En la tabla a continuación se puede observar los resultados. En Inbiomed, utilizando el kit Isopollo, obtuvimos una concordancia del 100 por ciento en la detección de CoVID19 en muestras de hisopado nasofaríngeo recibidas.
 |
Adicionalmente realizamos ensayos para conocer la eficiencia de los primers realizando diluciones seriadas con factor de dilución 1:2, esperando obtener 1 CT de diferencia entre cada dilución. El resultado esperado debería ser el incremento de 1 Cycle Threshold (CT) por cada dilución seriada, sin embargo, solo se cumplió esta condición entre la dilución 1:8 y 1:16. Las diferencias de CT en el resto de las diluciones no cumplieron con el requerimiento esperado. Estos resultados nos informaron que mientras más bajas son las concentraciones del control positivo, no existe una correlación directa que garantice una eficiencia coherente de los primers utilizados.
Conociendo las características del kit Isopollo, y una vez realizada la calibración y estandarización, decidimos testar el gen RpRd en todas las muestras recibidas en el laboratorio. Las muestras que resultaron positivas para este gen, fueron reportadas como diagnóstico de CoVID19. Y solo en el grupo de muestras negativas para el gen RpRd,se procedió a testar el gen N. Tomamos esta estrategia debido a que el kit Isopollo no es multiplex y debíamos duplicar la reacción y materiales necesarios para la determinación de los dos genes a la vez. Hemos, detectado el gen RpRd en el 99,5 por ciento de los pacientes positivos, confirmando la evidencia científica que reporta que este gen se encuentra en una región conservada del genoma viral, el cual posee bajos ratios de mutación. Los casos positivos para el gen N, también fueron reportados oportunamente como diagnóstico de CoVID19.
Compartimos esta aclaración técnica del procesamiento de los kits de PCR en tiempo real, para que se conozca el arduo trabajo que se realiza en los diferentes laboratorios que apoyan en el diagnóstico de CoVID19 y primordialmente para que la ciudadanía conozca que en el Inbiomed-UCE, lo realizamos con estándares profesionales de alto nivel.